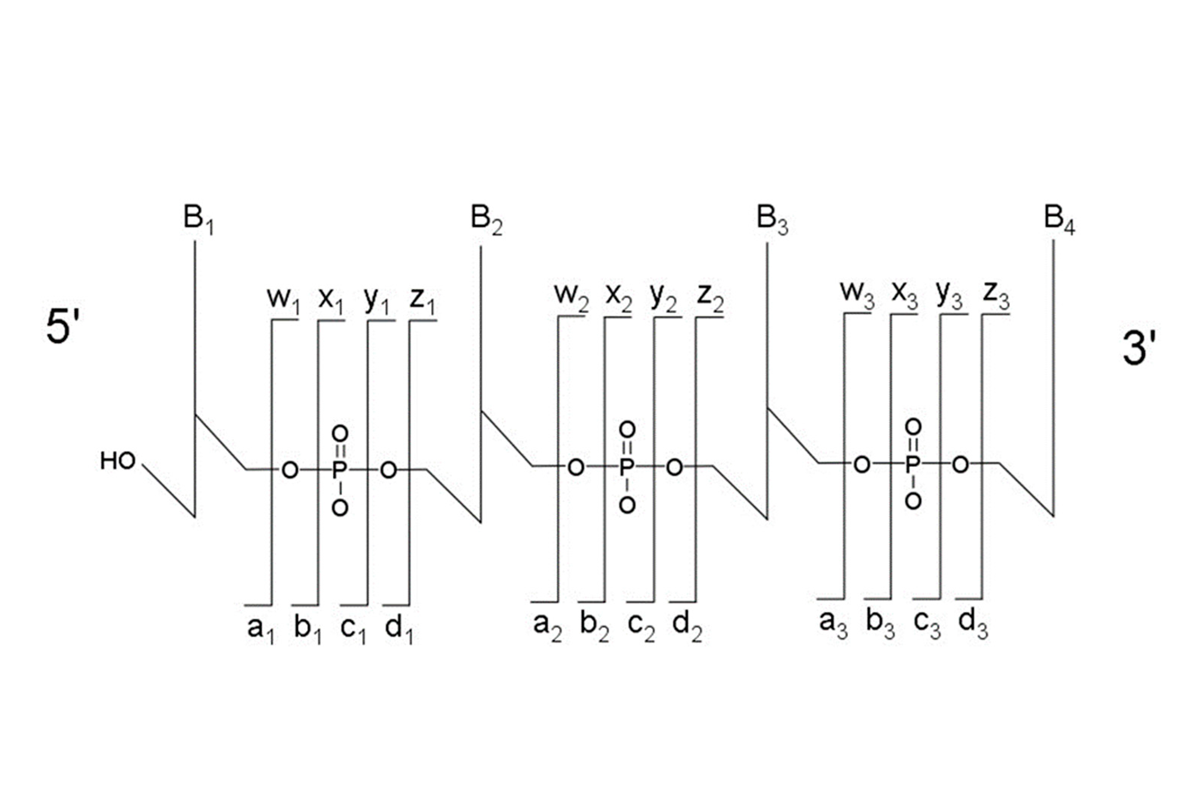
Resultados 1
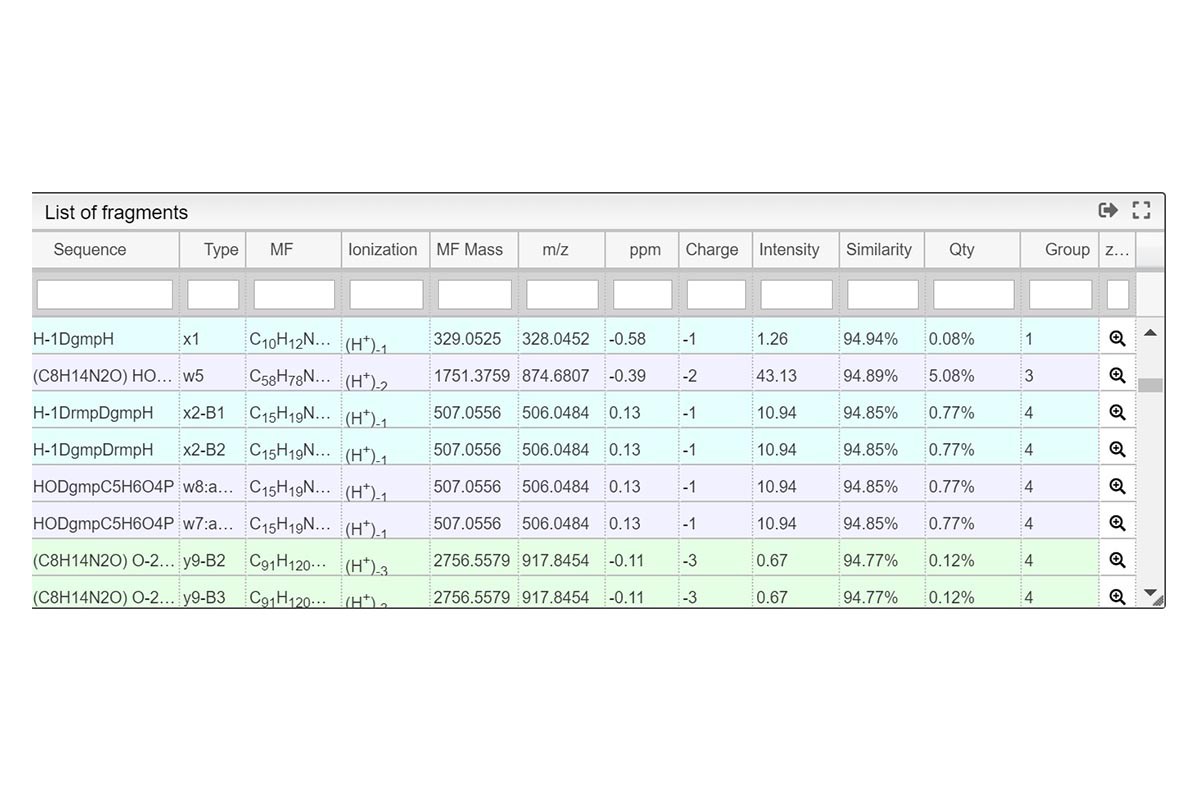
Vista de lista de fragmentos
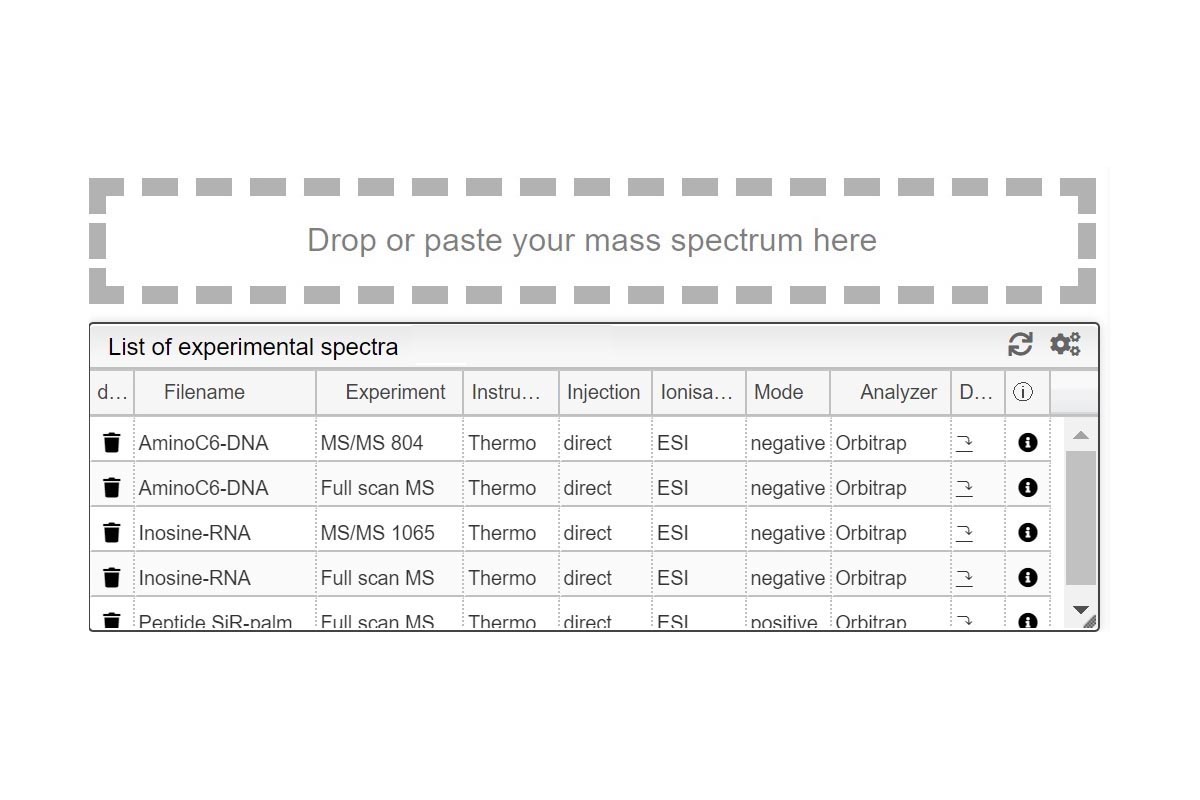
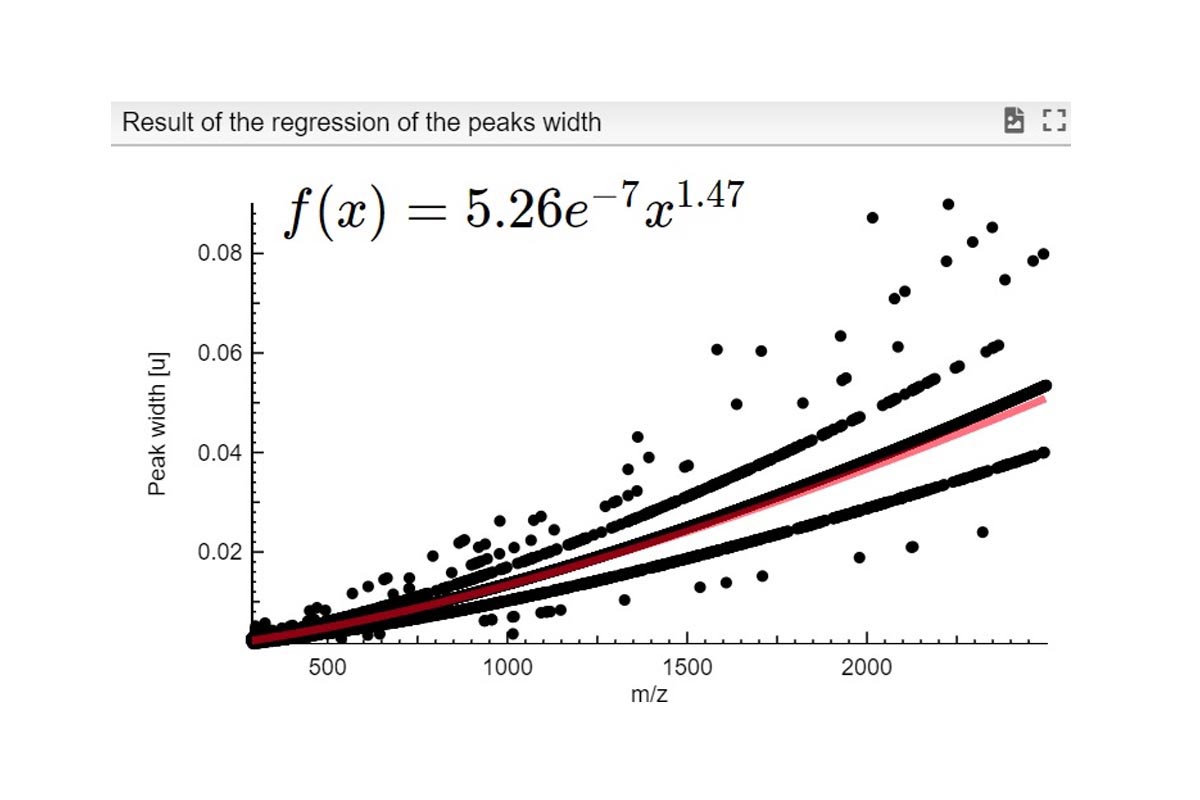
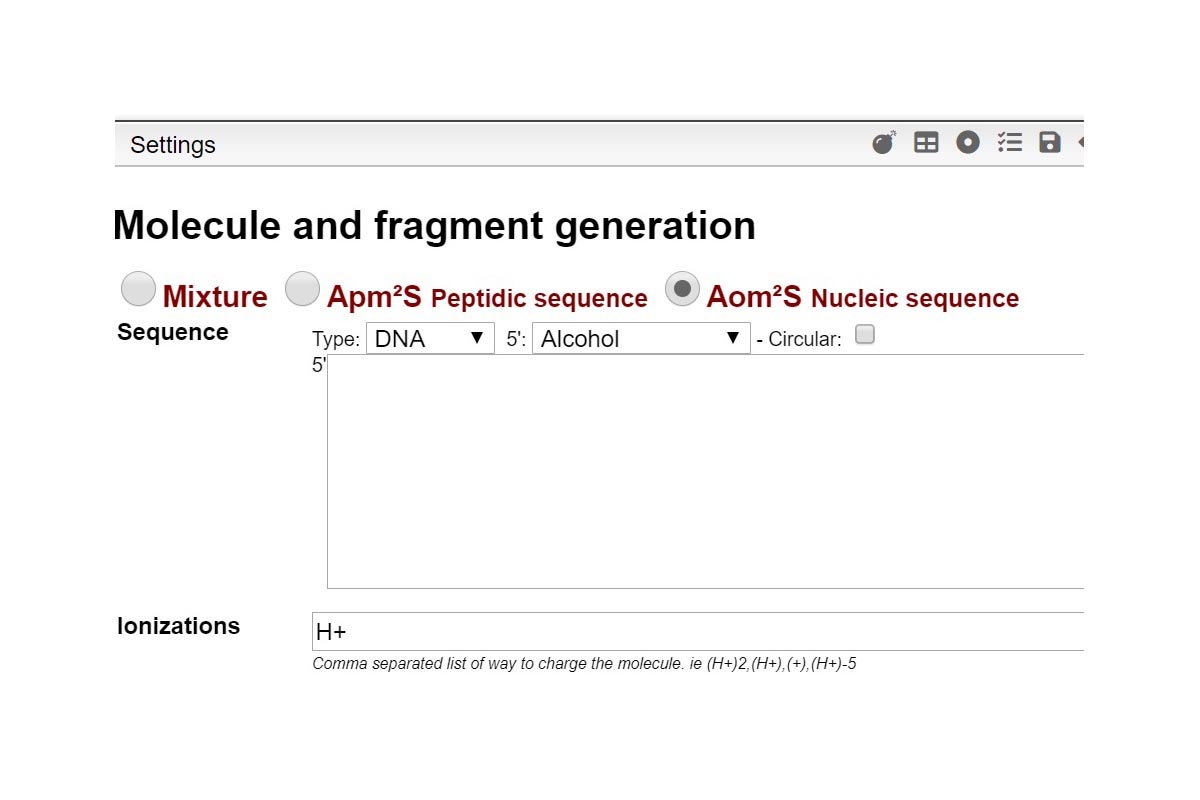
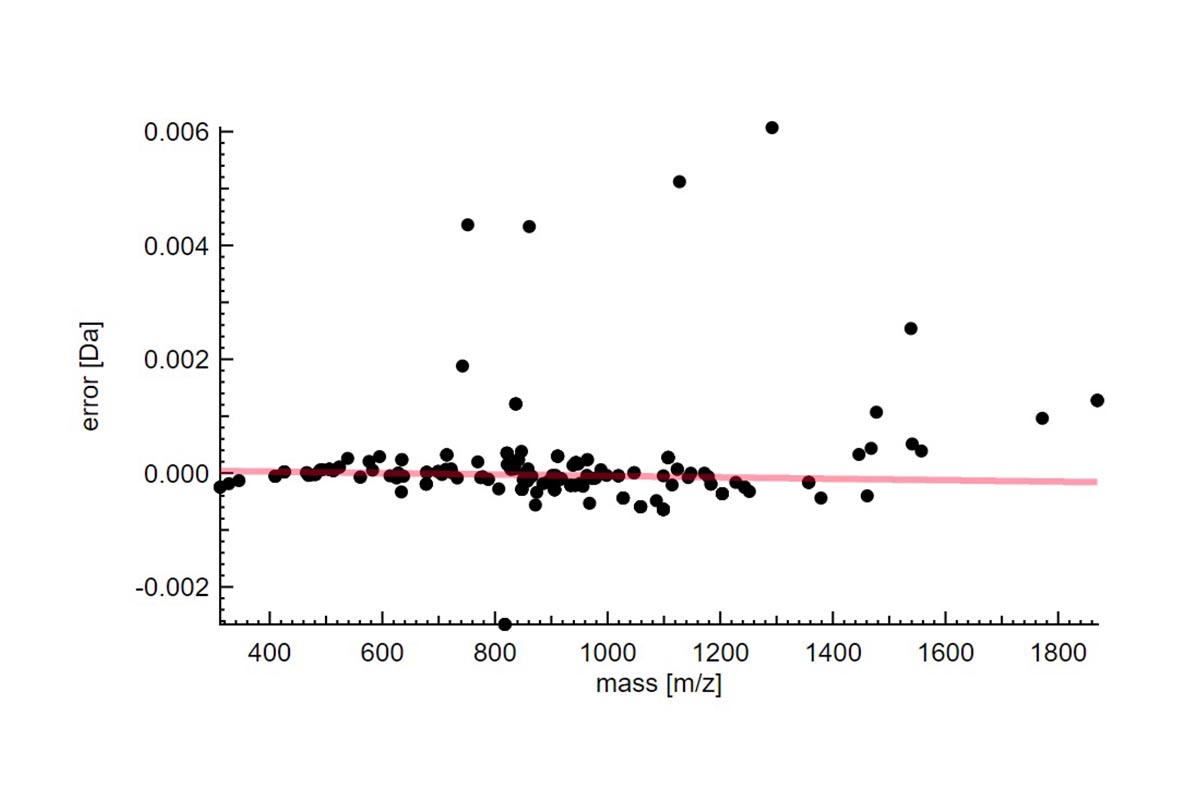
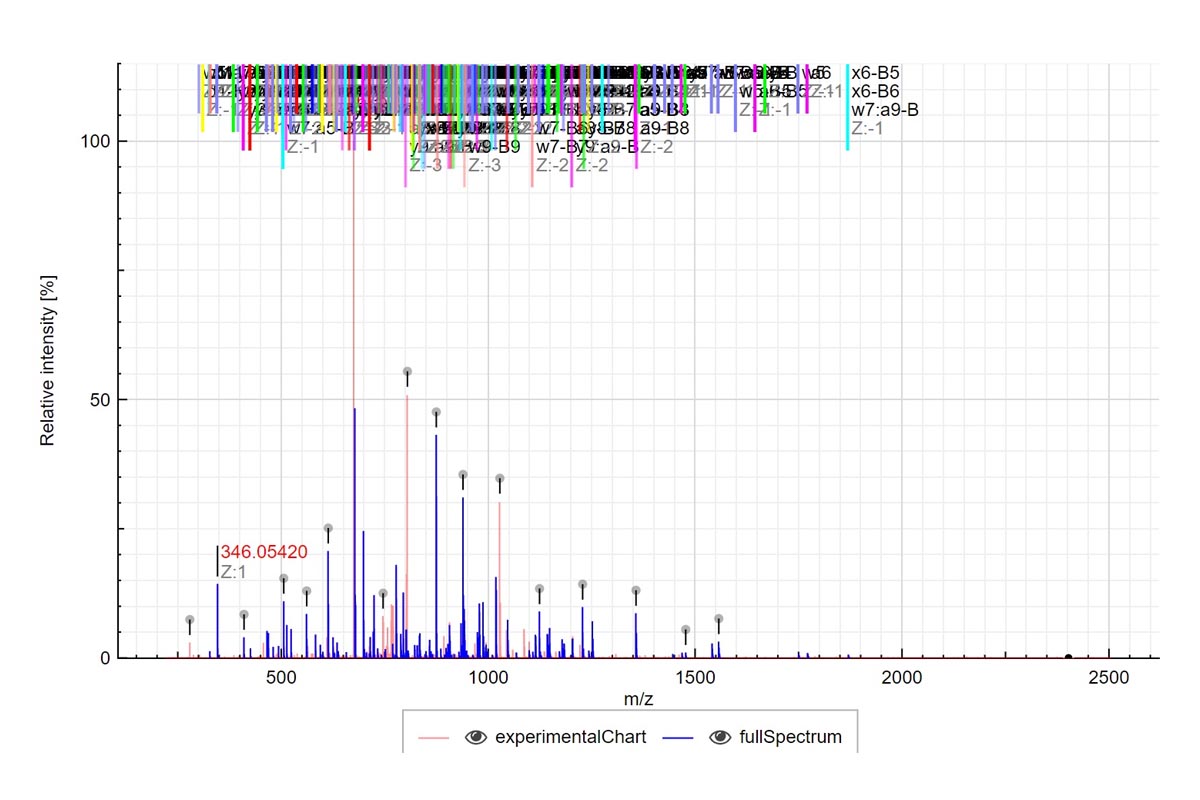
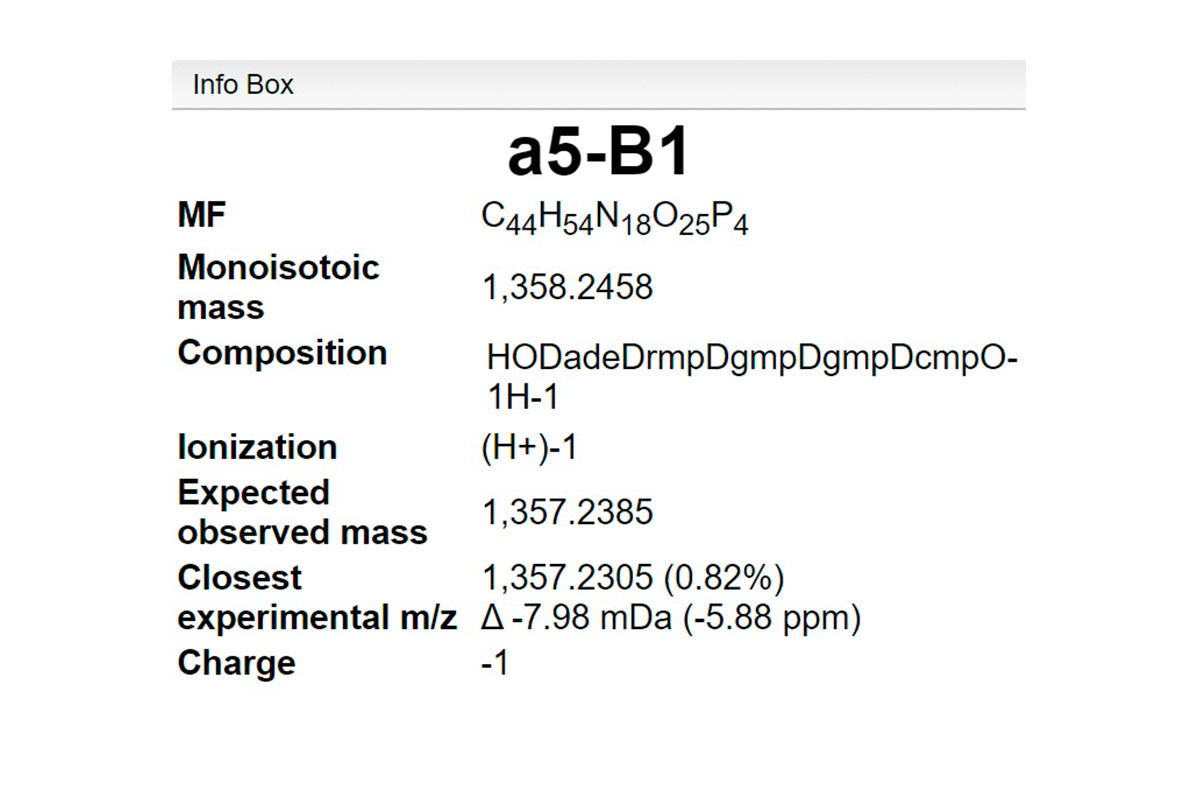
La primera vista es una tabla de resumen denominada “List of fragments” que contiene toda la información sobre las moléculas identificadas. Puede ver la fórmula molecular y la forma de ionización, así como el pico monoisotópico de la fórmula molecular (sin ionización) y la masa monoisotópica final incluida la carga. Otros valores interesantes como el error en ppm, la abundancia relativa, la cantidad, el grupo y el puntaje de similitud también se incluyen en esta vista. Para cualquiera de los fragmentos, puede hacer zoom e interactuar en la vista de espectros de masas (Resultados 2). Todos los datos presentados en la tabla se pueden clasificar por cualquier criterio con un simple clic en el encabezado de la columna. También puede usar la barra de búsqueda para buscar más rápidamente un valor individual o usarlo por rango (ir al glosario). También puede exportar toda la tabla como archivo de texto con el botón Exportar datos.