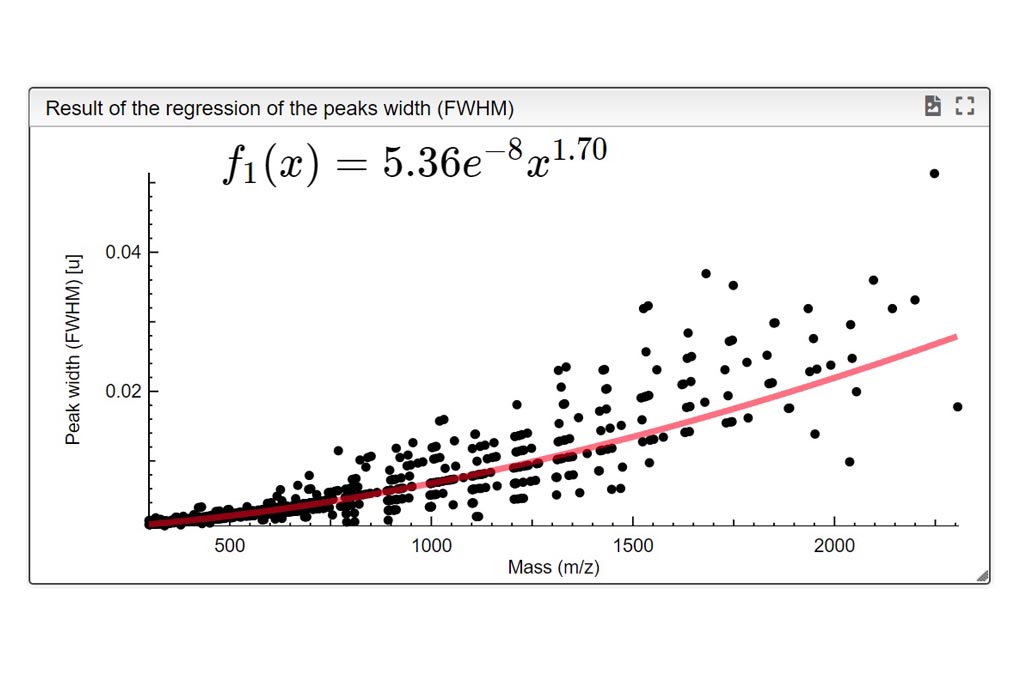
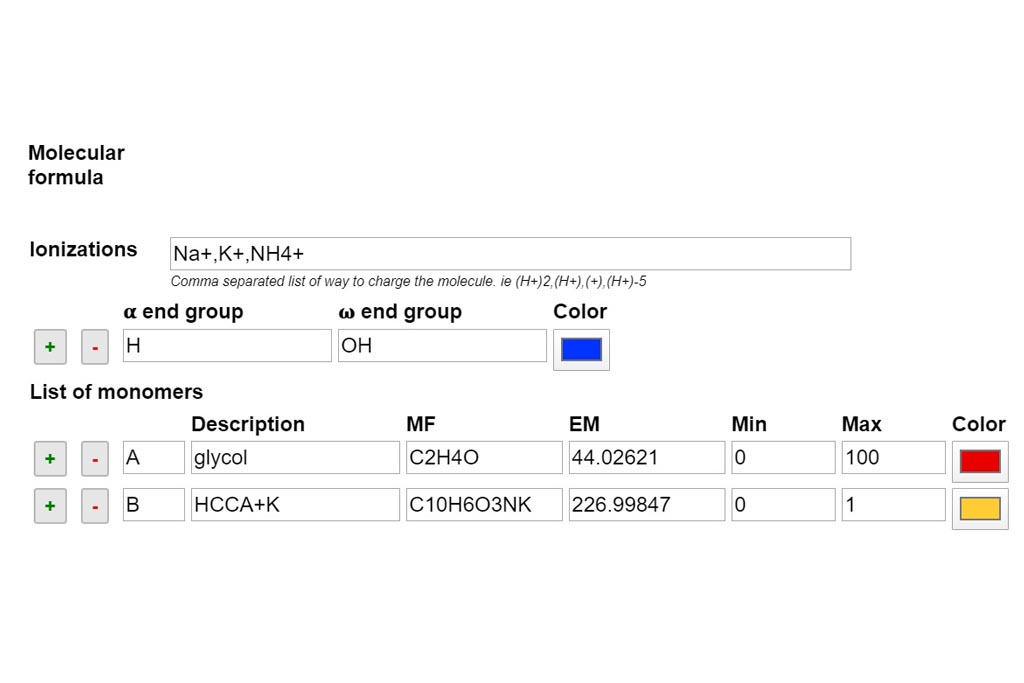
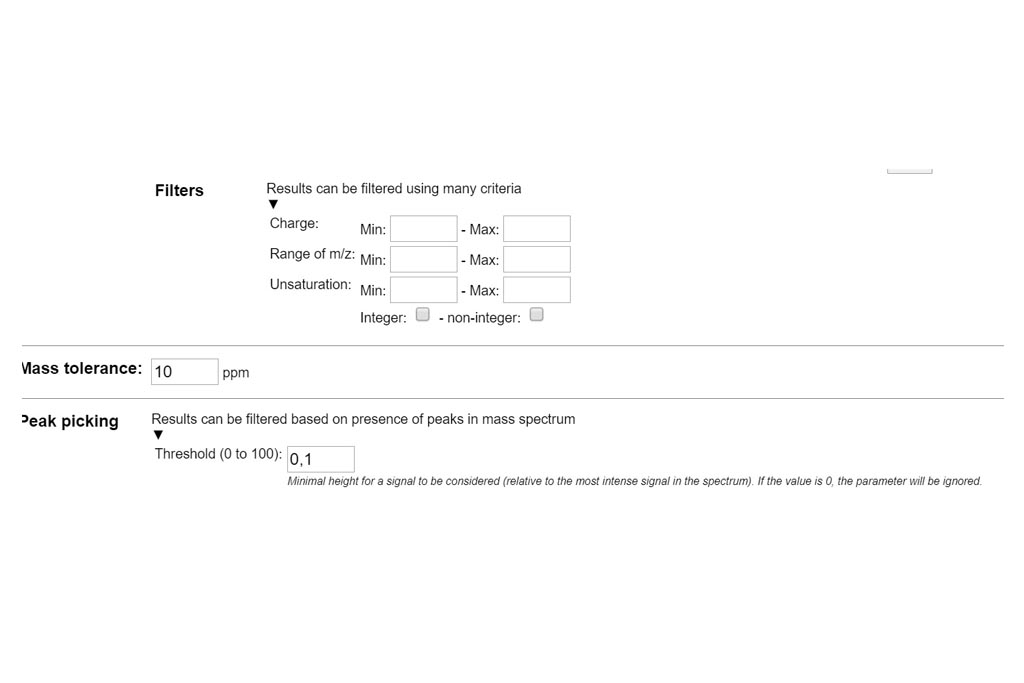
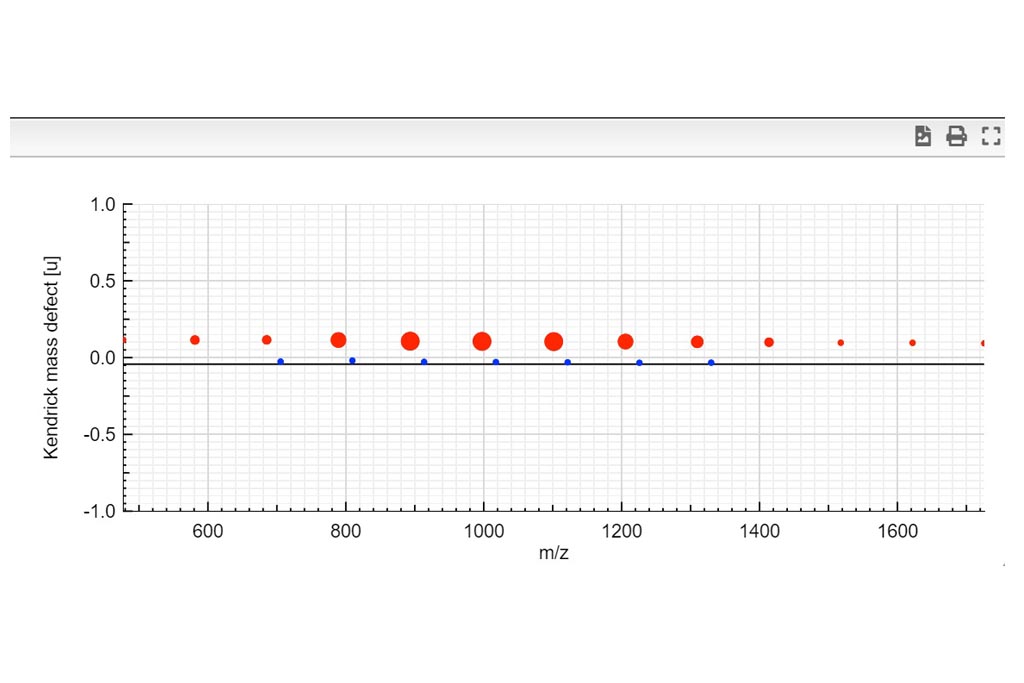
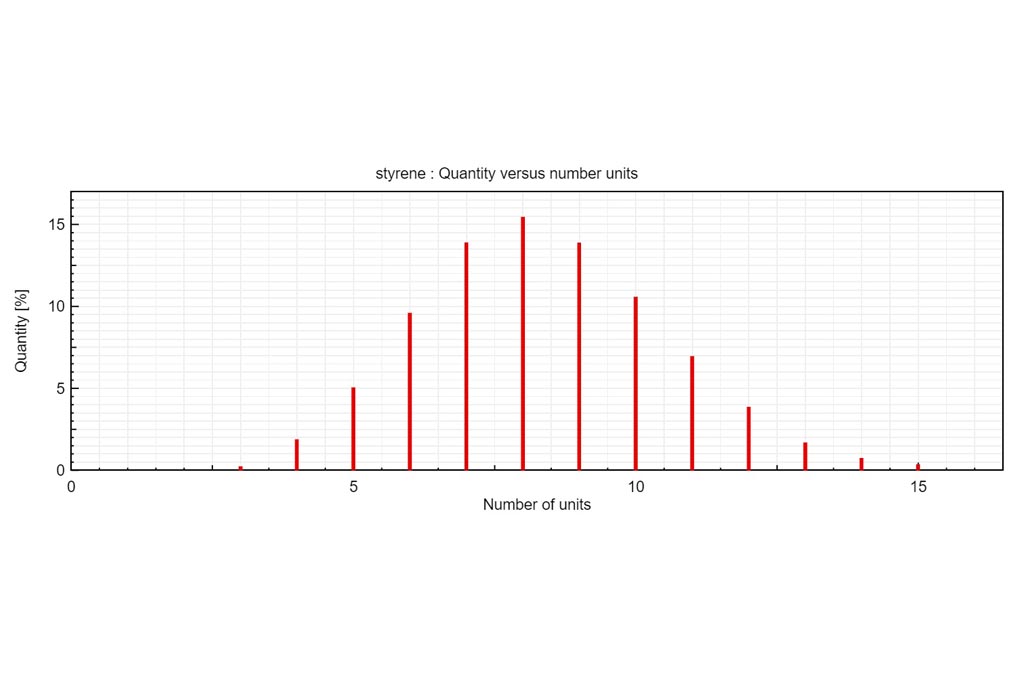
PASO 1
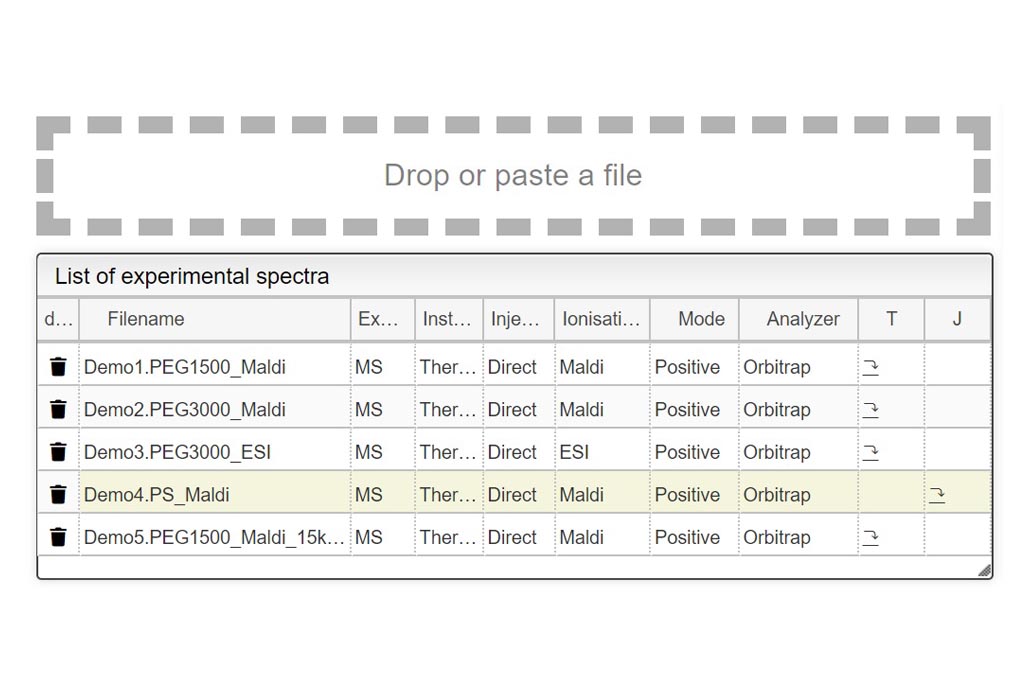
Importación de datos experimentales
Esta herramienta se puede utilizar en un LIMS o de forma independiente. En el modo independiente, debe arrastrar/soltar su espectro experimental como un archivo de texto delimitado por tabulaciones o copiarlo y pegarlo (CTRL-V) mientras mueve el mouse sobre la zona de colocación. La lista de espectros disponibles se mostrará en la tabla y simplemente puede hacer clic en el que desea mostrar.